

扫码打开虎嗅APP

本文经授权转载自微信公众号:BioArt(ID:BioGossip),作者:BioArt。
目前基因编辑有望纠正人类胚胎中的致病突变,但是包括CRISPR/Cas9和单碱基编辑在内的基因组编辑工具的临床应用需要在哺乳动物胚胎中进行无偏差的全基因组脱靶检测,但由于单核苷酸多态性等原因的存在,脱靶效应的程度难以确定。
11月27日,由中科院神经科学研究所、脑科学与智能技术卓越创新中心、中科院马普计算生物学研究所、斯坦福大学遗传学系等单位合作完成的研究论文Base editing generates substantial off-target single nucleotide variants在线提交了预印本杂志bioRxiv。在这项研究中,杨辉研究员等探索了全基因组范围内基因编辑技术可能造成的脱靶效应。
据BioArt了解到的消息,研究人员开发了一种名为GOTI的严格方法来检测脱靶效应,他们使用Ai9(CAG-LSL-tdTomato)基因工程公鼠与野生型C57母鼠杂交获得杂合基因工程受精卵,体外发育到二细胞阶段时,通过显微注射技术向一个卵裂球中混合注射Cre和CRISPR/Cas9或BE3基因编辑工具,从而编辑和标注一个卵裂球,随后比较发育到E14.5阶段的编辑和未编辑后代细胞的全基因组序列来进行全基因组脱靶分析,该方法考虑了不同个体之间的异质性,从而避免了不同个体遗传背景差异造成的影响。
他们利用该方法检测了CRISPR/Cas9和BE3编辑系统,发现 BE3编辑诱导了位于原癌基因(Pin1)和抑癌基因(Rsu1, Stk4, Tsc1, Park7, Rbpj, Ing3, Hipk2, Phlpp2, Yeasts4, Ptch1, Stk3, Smchd1)等基因上的数百个基因突变,这些脱靶性突变可能会引起肿瘤的发生,严重危害健康。
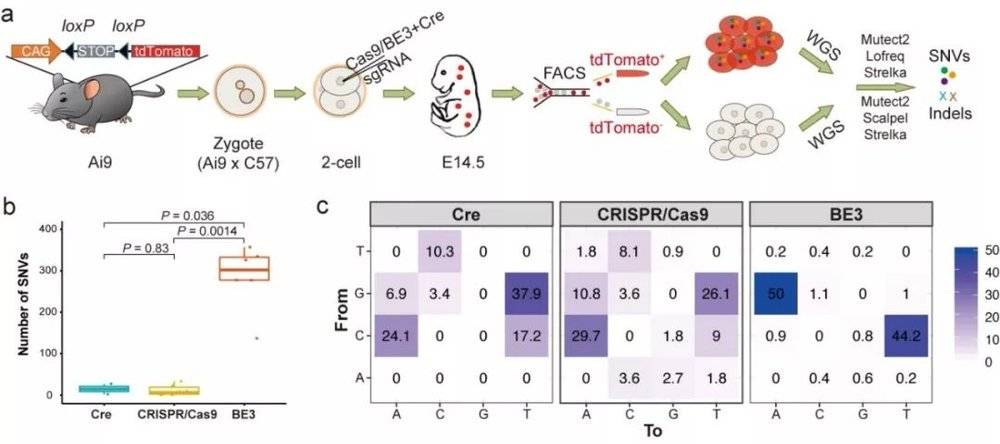
这项工作显示了基因编辑工具的安全性问题仍有待进一步评估和解决,也为未来CRISPR/Cas9的临床应用提供了一个可行的安全评估工具。
本文经授权转载自微信公众号:BioArt(ID:BioGossip),作者:BioArt。