

扫码打开虎嗅APP
本文来自公众号:机器之心(ID:almosthuman2014),作者: Synced
MIT 科学家用深度学习模型发现的“halicin”抗生素分子展示了前所未有的广谱抗菌能力,这是人类首次完全使用人工智能的方法发现新抗生素。研究人员表示,halicin 可以消灭一些世界上最危险的细菌。他们的这一研究登上了生命科学顶级期刊《Cell》。
自青霉素出现以来,抗生素已经成为现代医学的基石。然而,随着时间的推移,细菌会逐渐产生对抗生素的耐药性,这就需要药物研发工作者不断开发新的抗生素。
但现实情况是,由于缺乏经济激励,私营药企在新抗生素的发现上收效甚微,使得抗生素的问题越发严重。曾有研究预测,如果不立即采取措施开发新的抗生素,到 2050 年,因耐药感染而死亡的人数将达到每年 1000 万人。
在过去的几十年里,研发人员使用多种传统方法挖掘新的抗生素,但很多时候,他们会一次又一次地发现相同的分子,因此新抗生素的发现步履维艰。在此背景下,生物医学界亟需新的方法来帮助发现新抗生素。
解决 WHO“通缉令”上最危险的病原体
为了解决上述问题,来自 MIT 合成生物学中心的研究者开发了一种可以预测抗生素分子活性的深度学习方法,从超过 1.07 亿种分子中识别出了强大的新型抗生素分子——halicin。halicin 可以对抗多种细菌,如肺结核以及被认为无法治疗的菌株。而且,这种新发现的分子在结构上与已知的抗生素分子有很大不同。
在小鼠实验中,该分子对多种病原体具有抗菌活性,包括艰难梭菌(Clostridioides difficile)、肠杆菌科(Enterobacteriaceae)和有“广泛耐药性”并迫切需要新抗生素的鲍曼不动杆菌(Acinetobacter baumannii)。其中,后两者被世界卫生组织(WHO)列为高优先级病原体,是新抗生素三大研究目标中的两个。

虽然之前已有使用人工智能作用于部分抗生素发现的应用案例,但研究团队强调,此次最新发现是基于没有任何先前假设的情况下,完全从零开始识别出的全新抗生素种类。
目前,这项研究工作由麻省理工学院(MIT)的合成生物学家 Jim Collins 主导,已发表在权威学术期刊《Cell》上,并成为当期封面文章。

“就抗生素发现而言,这绝对是第一次,”该研究的作者之一、MIT 机器学习专家 Regina Barzilay 表示。
“我们希望能够开发一个平台,使人类能够利用人工智能的力量来开创抗生素药物发现的新时代。我认为 halicin 是迄今为止,人类发现的最强大抗生素之一,”MIT 团队生物工程师 James Collins 补充道。“它对于大量抗药性病原体展示了显著的活性。”
论文链接:https://www.cell.com/cell/fulltext/S0092-8674(20)30102-1
宾夕法尼亚州匹兹堡大学的计算生物学家 Jacob Durrant 表示,此项研究意义非凡,研究团队不仅确认了候选分子,同时还在动物实验中验证了有潜力的分子。更重要的是,这种方法具有一定的泛化性,可以应用于其他类型的药物研究,例如用于治疗癌症或者是神经退行性的疾病。
发现过程
在寻找新抗生素的过程中,研究者训练了一个深度神经网络,以寻找抑制大肠杆菌生长的分子。在训练过程中,他们用到了 2335 个已知具有抗菌活性的分子,包括大约 300 种已获批的抗生素和 800 种从植物、动物和微生物中得到的天然产物。
在这项研究中,模型不需要知道关于药物机理的假设,也不需要化学基团的标注就能执行预测。该模型能够学习人类专家未知的新模式。
训练完成后,研究者用该模型在一个名为“Drug Repurposing Hub”的库中进行分子筛选,里面有大约 6000 种正处于研究阶段的对抗人类疾病的药物分子。
此次筛选的目的是看哪种分子能对抗大肠杆菌,并挑选出那些看起来和常规抗生素不一样的分子。从这些结果里再选出 100 个候选分子进行物理试验,
结果,他们发现,一种名为“halicin”的分子效果很好,而这一分子正处于针对糖尿病治疗的研究阶段。在小鼠实验中,该分子对多种病原体具有抗菌活性,包括艰难梭菌、肠杆菌科和有“广泛耐药性”并迫切需要新抗生素的鲍曼不动杆菌。
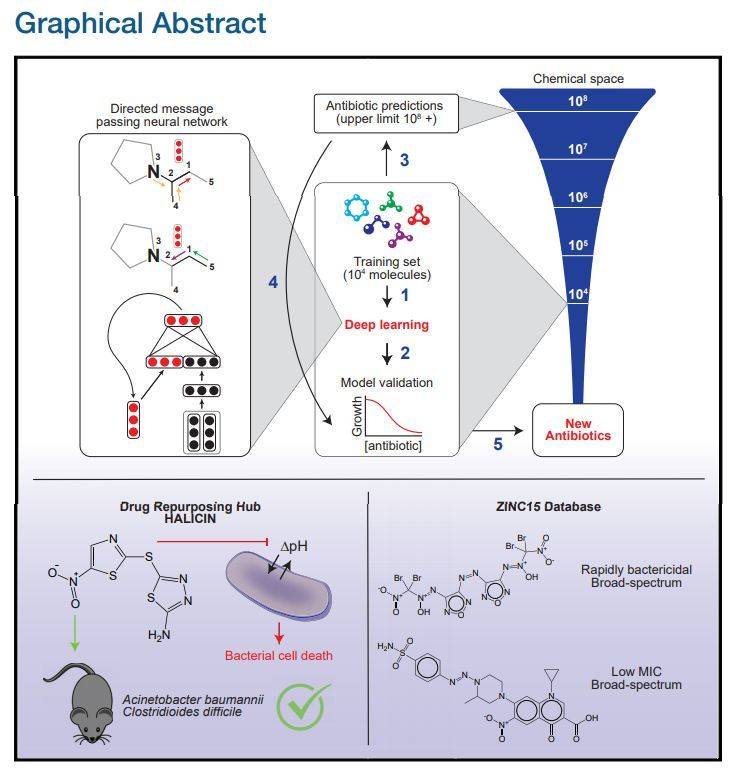
论文的图片摘要。
抗生素分子的图建模
整体来看,神经网络起到的作用就是预测不同分子结构最终对大肠杆菌的抑制能力。按照正常思维,我们需要一个数据集,其中输入特征是分子的组成部分与结构,标注为对大肠杆菌的抑制能力。而神经网络的优势在于,它能以一个自己学习的向量来表征分子,而不是手工设计的特征向量。
在这篇论文中,研究者表示有向信息传递网络能直接从分子的图结构预测分子的属性,其中原子可以表示为节点,化学键可以表示为边。对于每一个化学分子,研究者针对每一个组成部分的 SMILES 表达式建立分子图,其中 SMILES 是一种用 ASCII 字符串明确描述分子结构的规范。
在初始化特征向量时,它又分为原子特征向量与化学键特征向量。如下最左边的神经网络建模,其最上面为一个分子图,第 3 个原子和第 4 个原子都有连向第 2 个原子的化学键(Bond),下面两个黑色的向量表示这两个连接。以此类推,红色向量表示第 2 个原子连接到第 1 个原子的化学键。
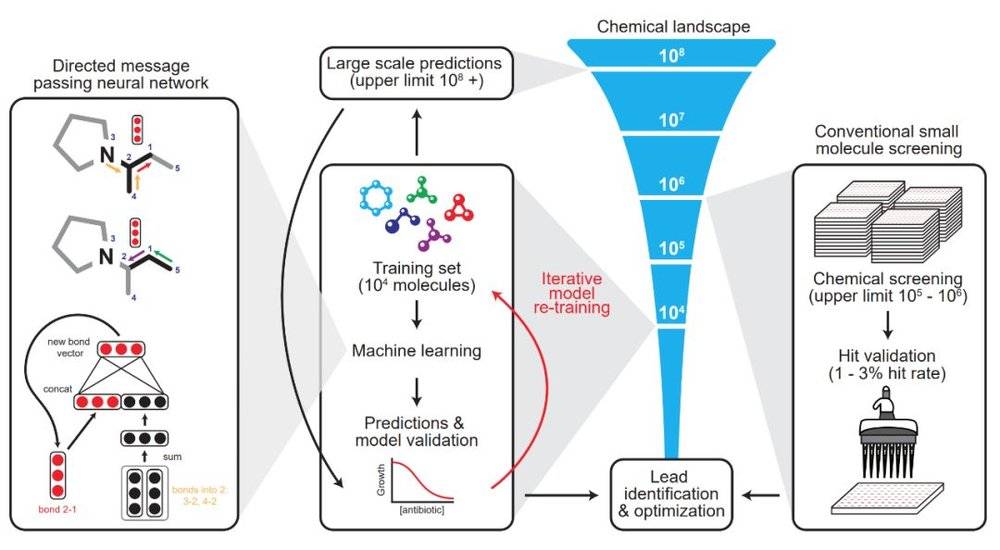
该模型应用了一系列信息传递步骤,它会聚合邻近原子与化学键的信息,从而理解局部分子的化学性质。在有向信息传递网络的每次信息传递过程中,通过求和邻近化学键特征向量,并馈送到非线性单层神经网络中,化学键特征向量能得到更新。
经过固定次数的信息传递步骤,整个分子学到的各种特征向量会加和为单个向量,并馈送到前馈神经网络以预测该化学分子对大肠杆菌的抑制效果。后面的 FFN 就是非常常规的二分类问题了,因此整个抗生素分子建模最重要的就是第一步的图模型:如何用图与向量表示分子之间的复杂关系。
这项开创性的工作标志着抗生素发现乃至更普遍的新药研发方法发生了范式转变。未来,MIT 的研究人员还计划使用深度学习模型来设计新的抗生素,并优化现有的分子。
其实,抗生素分子的发现只是机器学习在药物挖掘领域应用的冰山一角,全球各地的学者都在尝试用机器学习技术加快新药研发进度。以新型冠状病毒的新药研发为例,部分 AI 药物研发公司已经开始尝试用多种深度学习模型进行药物分子的挖掘(如英科智能利用二十多种模型寻找对新冠病毒关键蛋白酶有抑制作用的分子),相关成果也在陆续公布。
参考内容:
https://www.nature.com/articles/d41586-020-00018-3
http://news.mit.edu/2020/artificial-intelligence-identifies-new-antibiotic-0220
https://www.theguardian.com/society/2020/feb/20/antibiotic-that-kills-drug-resistant-bacteria-discovered-through-ai
https://insilico.com/ncov-sprint
本文来自公众号:机器之心(ID:almosthuman2014),作者: Synced
